上海吉玛制药技术有限公司
18 年
手机商铺
商家活跃:
产品热度:
- NaN
- 0
- 0
- 1
- 0
推荐产品
公司新闻/正文
mitoRNAi 现象为线粒体基因功能研究带来新机遇
2647 人阅读发布时间:2021-04-06 18:10
小 RNA(small RNA),包括小干扰 RNA(siRNA)、microRNA(miRNA)和 piRNA 在真核细胞基因表达调控中起着关键作用。RNA 干扰(RNA interference, RNAi)技术已经被广泛用于研究不同生物学背景下的基因功能。 有趣的是,迄今为止人们还不清楚核糖核酸酶样的机制是否可以在线粒体中发挥作用。已知 Argonaute 是 RNA 诱导沉默复合物(RISC)中的一种关键酶,负责高等真核细胞中的靶向 RNA 降解,由于 miRNAs 和 siRNAs 基本上利用相同的机制来处理和发挥作用,已经证明,在线粒体中特定的 miRNAs 不仅可以检测到,而且在线粒体中也有功能,问题在线粒体中是否也有类似 RNAi 的机制发挥作用。
线粒体是真核细胞中的内共生细胞器,可作为通过氧化磷酸化(OXPHOS)产生 ATP 的强大动力。线粒体基因组在进化过程中变化最小。如人类线粒体基因组的长度仅约 16kb,编码 2 个必需的 rRNA 和最少的 22 个 tRNA 进行翻译,以及 13 个多肽,所有这些都是膜蛋白,是呼吸链复合体的一部分,线粒体中的所有生化活动都需要核基因组(nDNA)编码的蛋白质。但已知针对线粒体中相关基因调控的手段和方法相对匮乏。
2020 年 8 月,加州大学圣地亚哥分校付向东及中国科学院生物物理研究所张晓荣共同通讯在 Cell Research 在线发表题为「Active RNA interference in mitochondria」的研究论文,该研究报道转染的 siRNA 不仅能够进入线粒体基质,而且还可以在那里特异性沉默靶向的线粒体转录本。
首先作者通过转染 Cy3 标记 21nt 的 siRNA 进入 C2C12 细胞。再通过线粒体基质蛋白(即 HSP60 和 MRPL44)富集出转染细胞线粒体,在缺乏 Triton X-100 的条件下通过胰蛋白酶处理去除特定外膜(即 Tom20)和内质网(即 ERP44)蛋白质,这样可以得到高纯度线粒体 (Fig. 1a).。对提纯的线粒体中含转染的荧光 siRNA 进行 RNA 酶消化处理,发现 siRNA 在没有 Triton X-100 的情况下(备注:Triton-100 有膜通透性),对 RNase T1 加微球菌核酸酶(microoccal,MNase)处理具有抗性,并且在 Triton X-100 的存在下对这些核酸酶变得敏感 (Fig. 1b)。此外,作者为了观察转染的 siRNA 是否在线粒体中定位分布,我们用 FAM 标记的 COXI siRNA 转染到含共表达表达 TOM20 mCherry(标记外膜)和 su9-BFP(标记线粒体基质的 F0-ATPase 亚基 9 的线粒体前序列)的小鼠胚胎成纤维细胞(MEF)细胞。位于 su9-BFP 标记小室的 FAM 信号, 表明 siRNA 在线粒体基质中的分布(见论文补充信息,图 S1 和视频 S1)。这些发现为外源性 siRNA 在线粒体基质中定位的能力提供了有利证据。该课题组进步系统地研究 siRNA 和小发夹 RNA(shRNA)是否能够靶向 mtDNA 编码的转录本。发现随机设计的 siRNA(scramble siRNA)并未显示出预期的干扰效果,但根据 Argonaute 2(Ago2)结合原理设计的特异性 siRNA 都能够有效的以 Ago2 依赖性的方式靶向 mtDNA 编码的转录本发挥预期的干扰效果(Fig 2)。但同时由于大多数线粒体蛋白都非常稳定,从而阻止了在蛋白水平上检测到预期的 RNAi 效应,这可能解释迄今为止缺乏关于活性 mitoRNAi 的报道,另外 small RAN 进入线粒体的机制还有待挖掘。
综上所述,尽管 small RNA 进入线粒体的机制尚不清楚,但 siRNA 和 miRNA 进入线粒体内进行调控 mtDNA 编码基因的功能将是非常有用的工具,也有证实,miRNA 可以增强的线粒体翻译功能,如 miR-21 调控线粒体转录表达在大鼠自发性高血压模型中降低血压的作用有关,这为证明的线粒体中 miRNA 干扰可提供一种由线粒体基因组突变引起疾病的潜在治疗性干预策略。因此,新建立的 mitoRNAi 有望推动线粒体生物学的基础和临床研究。

Fig. 1: Detection of small RNAs in mitochondria by nuclease protection, in vitro RNA import and ClickIn assays.
a The purity of isolated mitochondria analyzed by western blotting for HSP60 and MRPL44 (mitochondrial matrix proteins), Tom20 (mitochondrial outer membrane protein), and ERP44 (endoplasmic reticulum protein). b The distribution of Cy3-labeled siRNA 4 h after transfection into C2C12 cells. Purified mitochondria were subjected to the nuclease protection assay with RNase T1 plus MNase. RNAs were resolved by urea or agarose gel and developed by Typhoon scanning or EtBr staining. c 32P- or Cy3-labeled RNA molecules of indicated lengths were analyzed by nuclease protection assay after incubation with purified mouse heart mitochondria. d Schematic diagram of the ClickIn assay. MitoOct carries a PPh3 group with high affinity to mitochondrial matrix and a cyclooctyne (Oct) group reactive to azide. FCCP, carbonyl cyanide 4-(trifluoromethoxy) phenylhydrazone; Tet, tetrazine. e Mitochondria purified from mouse heart were incubated with 32P-labeled and azide-labeled siCOXI followed by the addition of MitoOct in the presence or absence of FCCP. The upper band indicates the MitoOct-modified siRNA in mitochondrial matrix. T1, RNase T1; MN, micrococcal nuclease; MT, mitochondria.
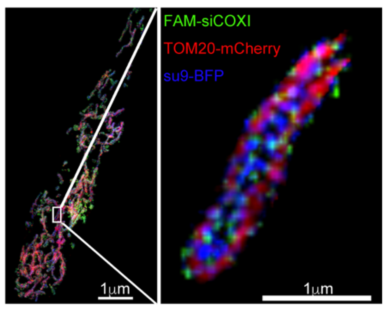
图 S1 和视频 S1
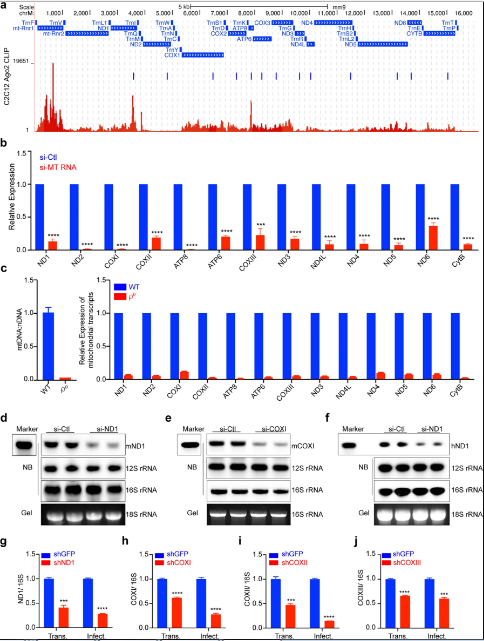
Fig. 2: Specific targeting of mtDNA-encoded transcripts by siRNA and shRNA.
a siRNAs designed to target individual mtDNA-encoded transcripts according to previously mapped Ago2-binding peaks in C2C12 cells. b RT-qPCR analysis of all 13 mitochondrial transcripts in C2C12 cells after treatment with corresponding siRNAs. c qPCR analysis of mtDNA copy numbers in ρ0 and WT C2C12 cells (left panel); RT-qPCR analysis of transcripts in ρ0 and WT C2C12 cells (right panel). d–f Northern blot (NB) analysis of mouse ND1 and COXI mRNAs in C2C12 cells (d, e) and human ND1 mRNAs in HeLa cells (f) treated with either control or specific siRNAs. rRNAs serve as loading control and were detected by northern blotting or direct SYBRTM Gold staining in the same gel. g–j RT-qPCR analysis of COXI, II, III, and ND1 RNA levels in HEK293T cells transiently transfected (Trans.) or stably expressed (Infect.) shCOXI, shCOXII, shCOXIII, and shND1. Data represent means ± SEM based on three independent experiments. ***P < 0.005, ****P < 0.001 based on unpaired Student’s t-test.
参考文献:
Gao, K., Cheng, M., Zuo, X. et al. Active RNA interference in mitochondria. Cell Res (2020). https://doi.org/10.1038/s41422-020-00394-5
目前公司拥有处于国际先进水平的 siRNA 化学合成的全部核心技术,包括 RNA 单体合成技术、普通和修饰的 siRNA oligo 合成技术、核酸荧光标记技术、多种核苷酸化学修饰技术、shRNA 质粒载体构建技术,慢病毒载体构建以及包装技术、microRNA 荧光定量 PCR 检测试剂盒、荧光定量 PCR 探针合成技术及其荧光定量 PCR 检测技术等。在基因编辑技术上,公司拥有化学合成 gRNA、载体构建、病毒包装等多种平台,结合载体构建及直接使用 Cas-9 蛋白等方法实现基因编辑。在此基础上,吉玛公司已经发展形成强大的细胞技术平台,包括细胞增殖检测 (MTT & CCK-8 )、细胞凋亡检测(Annexin V/PI)、细胞迁移 & 侵袭实验 (Transwell)、酶联免疫吸附实验(ELISA)等。此外,公司也建立了规范的 SPF 级动物房,开展转基因动物、PDX 模型建立和应用等平台。在诊断试剂开发上已经拥有一个近千平米的三类诊断试剂 GMP 生产车间。展望未来:RNAi 技术和基因编辑技术在充满挑战的后基因组时代必将有广阔的应用前景,将会在生物医学研究中产生一种新的技术革命。化学合成的 siRNA、 miRNA、反义核酸、核酸适配体,化学合成的 gRNA 等基因药物的开发将成为极具发展前途的新兴产业。

公司外貌

高通量合成仪


